卢艳丽教授团队开发出预测植物蛋白质点突变功能效应的机器学习工具PPVED
作者: 审稿人:ymyjs 时间: 2022-04-13 点击次数:次
近日,玉米研究所卢艳丽教授团队在植物学知名期刊《Plant Biotechnology Journal》在线发表了一篇题为“PPVED: A machine learning tool for predicting the effect of single amino acid substitution on protein function in plants”的研究论文,该研究论文开发了一个机器学习工具PPVED(网站链接为http://www.ppved.org.cn),实现了植物蛋白质点突变功能效应的准确预测,极大地促进了植物功能性遗传变异的识别与挖掘。玉米研究所硕士毕业生苟香建(现为华中农业大学植物科学技术学院博士一年级学生)为第一作者,青年教师冯宣军为共同第一作者,卢艳丽教授为通讯作者。

蛋白质点突变,即单氨基酸替换(Single Amino Acid Substitution,SAAS),可影响蛋白质行使正常功能,从而导致植物发生明显的生理或形态改变。传统的分子实验能够准确衡量SAAS对植物蛋白质功能的影响;然而,这些实验的开展非常耗时耗力且操作困难。大量全基因组重测序项目的实施也导致了SAAS数目急剧增加,传统实验验证SAAS变得更加低效。因此,为了从海量数据中筛选出最有价值的SAAS,优先进行分子实验验证,急需一种能够大规模、高通量、高精度注释SAAS效应的新方法。
该团队利用UniProt/Swiss-Prot、NCBI/PubMed等数据库资源,整合了一个植物蛋白质点突变数据库,一共包含12,865个SAAS,并将其随机划分为基准数据集和独立数据集。基于基准数据集,从蛋白质序列、结构与功能等层面出发,广泛采集了1,215种表征SAAS的特征信息,使用自行设计的特征选择管道筛选出48种重要的特征。基于这48种特征,分别应用随机森林、极端梯度提升、支持向量机以及前馈神经网络等算法,构建了15种机器学习模型。评估结果表明,以极端梯度提升算法构建的模型(命名为植物蛋白质变异效应检测器,Plant Protein Variation Effect Detector,PPVED)性能最优,在基准数据集上的预测准确度和曲线下面积分别为0.872和0.940,在独立数据集上分别为0.856和0.931。

图1.关于开发PPVED并用其预测SAAS对植物蛋白质功能影响的概述
该团队从三种角度进一步验证了PPVED的预测能力。首先,比较了PPVED与6个广受欢迎的现有工具(基于人类SAAS数据而开发)的性能,结果表明,PPVED的预测准确度比性能最优的现有工具高出10%。其次,将PPVED预测应用于包含314个功能性SAAS和1,515个中性SAAS的数据集,结果表明,预测准确度与独立数据集的预测结果一致,再次证实了PPVED的泛化能力。最后,利用PPVED对拟南芥、玉米中的6个SAAS(ISU1T55M、ISU1A143T、ISU1G106D、HSCA2G87D、ZmERF9T211S、ZmERF9R204Q)进行预测和实验验证。结果表明,除了ISU1T55M之外,对其他5个SAAS的预测与实际观测结果保持一致。
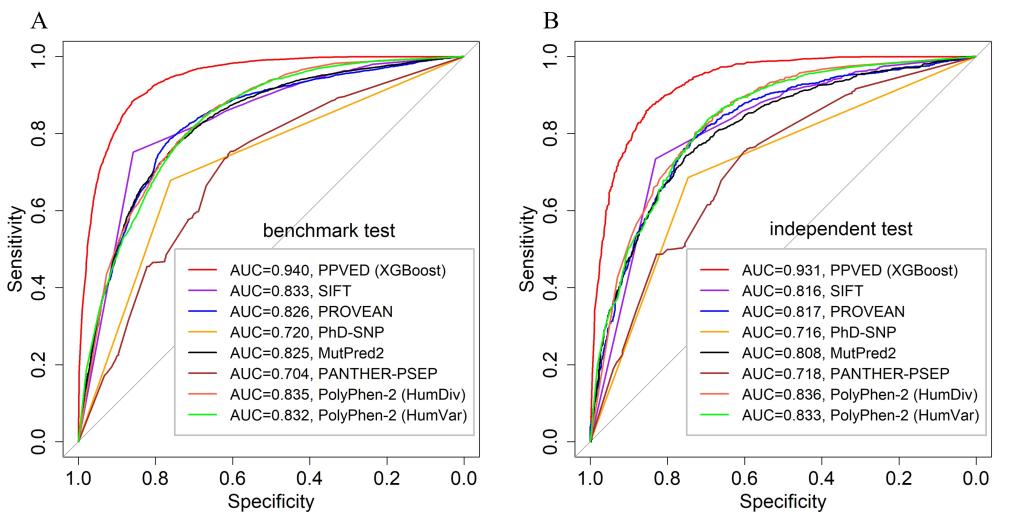
图2. PPVED与6个现有工具在基准数据集和独立数据集中的ROC曲线
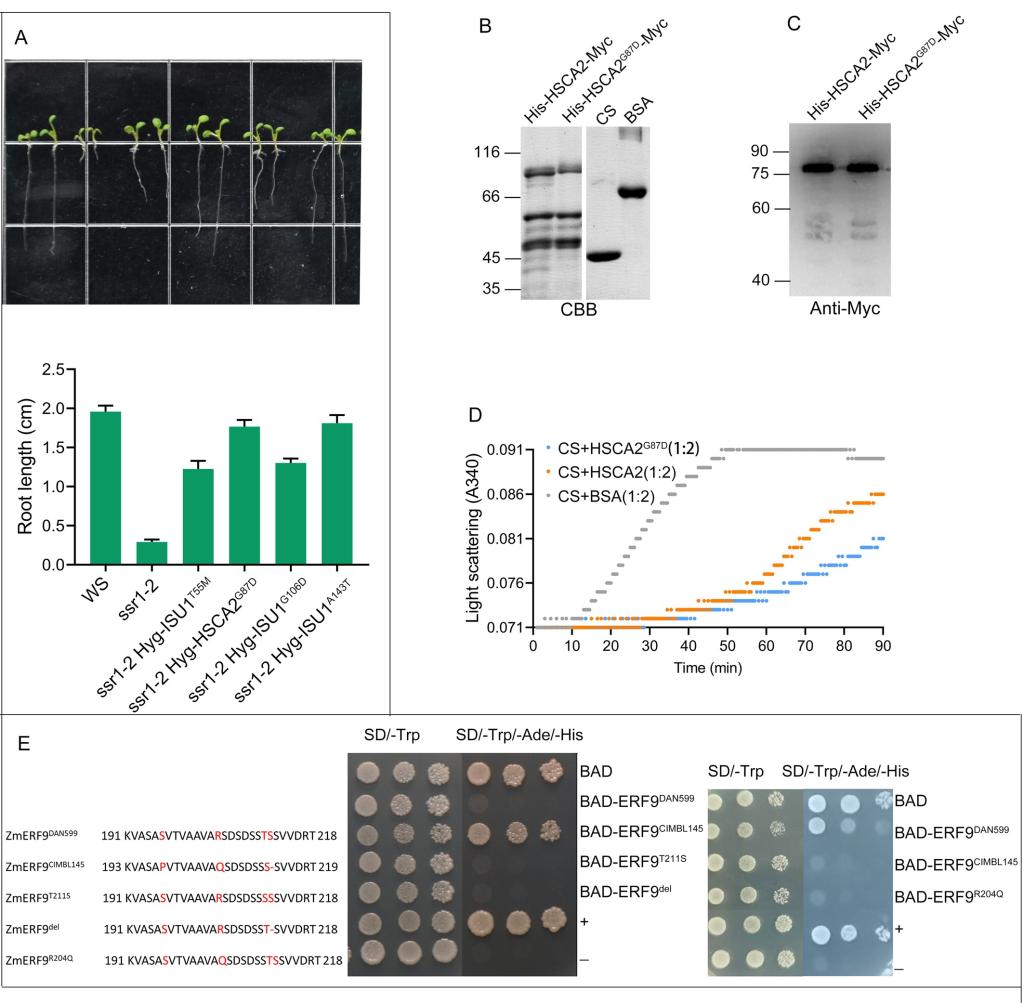
图3.拟南芥与玉米中的6个SAAS的验证结果
为了方便用户使用PPVED,作者将PPVED部署成了在线Web服务,用户可以直接登录网站,提交任务并进行预测,结果会以邮件的形式发给用户的邮箱,网站链接为http://www.ppved.org.cn。此外,该网站也提供了本地安装包,适用于64位的Linux操作系统,如果用户有批量预测需求,作者推荐用户下载本地安装包进行批量计算。

图4. PPVED的在线Web服务界面
供稿人:冯宣军
审稿人:高世斌
论文链接:https://onlinelibrary.wiley.com/doi/abs/10.1111/pbi.13823


